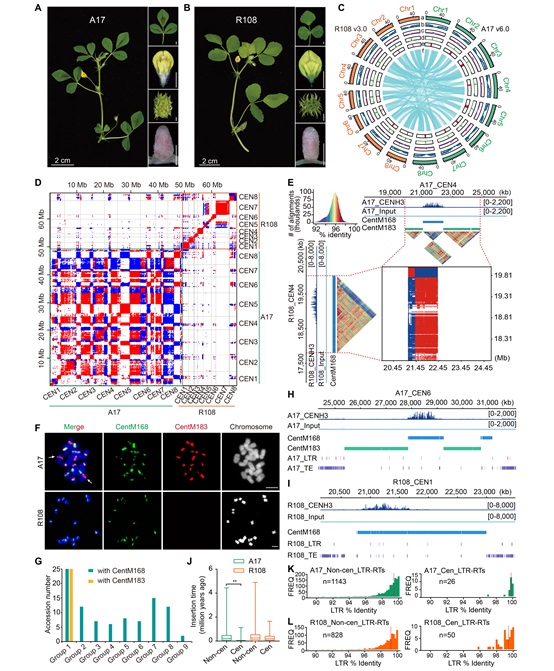
苜蓿(Medicago)是重要的豆科模式植物,也是典型的高蛋白饲草作物。其根系能与土壤中的根瘤菌互作共生、结瘤固氮,从而促进植物生长和土壤肥力的提升。深入解析苜蓿基因组的结构、功能和演化机制,挖掘有重大育种价值的新基因,将为提高苜蓿产量、结瘤固氮能力和环境韧性奠定分子基础,有助于指导创制新型苜蓿种质。近日,中国科学院遗传与发育生物学研究所冯健研究团队与韩方普研究团队合作,成功完成了两个代表性苜蓿材料——Medicago truncatula Jemalong A17 (以下简称为A17) 和 Medicago littoralis R108 的端粒到端粒(telomere-to-telomere, T2T)无缺口基因组组装,并首次系统解析了其着丝粒区域的结构组成及演化特征。相关成果以“Two complete telomere-to-telomere Medicago genomes reveal the landscape and evolution of centromeres”为题,于2025年7月25在线发表于国际植物学权威期刊《Molecular Plant》。
在该研究中,团队综合应用PacBio HiFi高精度长读长测序、Oxford Nanopore超长读长测序及Hi-C三维基因组构象捕获技术,分别构建了A17和R108材料的多套组装方案,最终获得了高质量、无缺口的T2T基因组版本:A17 v6.0和R108 v3.0。A17基因组为494.47 Mb,R108基因组为415.27 Mb,BUSCO完整性评估均超过99%,全部染色体实现了真正意义上的端粒到端粒连续组装,是目前苜蓿属植物最完整的参考基因组资源。
在此基础上,研究团队重点关注着丝粒(centromere)区域的结构特征及演化规律。通过结合CENH3染色体免疫共沉淀、荧光原位杂交和重复序列注释分析等数据,发现A17与R108材料的着丝粒结构具有显著差异:A17的着丝粒主要由两类特异性卫星重复序列CentM168和CentM183组成,其中CentM183为M. truncatula所特有;而R108的着丝粒则几乎完全由CentM168构成,体现了其来源物种M. littoralis的独特演化轨迹。这一发现揭示了不同种苜蓿植物的着丝粒快速分化过程及卫星重复序列的动态变化规律,并提出了一个新的着丝粒结构稳定机制:活性着丝粒区域由CENH3富集的主要卫星序列CentM168构成,而非活性区域则由其他卫星重复序列环绕保护,从而有助于避免染色体重排或断裂。此外,该研究还鉴定出多个特异性卫星重复序列(例如CentM51、CentM515、CentM287等),在不同染色体上呈现高度位点专一性,进一步印证了苜蓿着丝粒的快速演化特征。值得注意的是,该研究还发现着丝粒区域的LTR反转录子插入更为活跃,且其序列相对较为年轻,暗示这些元件在苜蓿着丝粒的形成与维持过程中可能发挥关键作用。总体而言,该研究不仅构建了苜蓿属两个代表性材料的最完整参考基因组资源,也为豆科植物基因组研究、重要功能基因挖掘和着丝粒演化机制探索提供了坚实基础和关键支撑。
中国科学院遗传与发育生物学所冯健研究员和韩方普研究员为本文共同通讯作者。冯健研究组申莉莎助理研究员、韩方普研究组博士后易从杨和副研究员刘阳为该文章共同第一作者。该研究得到了国家科技重大专项和国家自然科学基金的资助。